

updated on 2019-08-18

ニューラルネットワークの性能の“悪さ”を示す指標で一般には、2 乗和誤差や交差エントロピー誤差などが用いられる
def mean_squared_error(y, t): return 0.5 * np.sum((y-t)**2) >>> # 「2」を正解とする >>> t = [0, 0, 1, 0, 0, 0, 0, 0, 0, 0] >>> >>> # 例 1:「2」の確率が最も高い場合(0.6) >>> y = [0.1, 0.05, 0.6, 0.0, 0.05, 0.1, 0.0, 0.1, 0.0, 0.0] >>> mean_squared_error(np.array(y), np.array(t))0.097500000000000031
def cross_entropy_error(y, t): delta = 1e-7 return -np.sum(t * np.log(y + delta))
np.log(0) はマイナスの無限大を表す-inf となり、そ うなってしまうと、それ以上計算を進めることができなくなります。その防止策とし て、微小な値を追加して、マイナス無限大を発生させないようにしています。
>>> t = [0, 0, 1, 0, 0, 0, 0, 0, 0, 0] >>> y = [0.1, 0.05, 0.6, 0.0, 0.05, 0.1, 0.0, 0.1, 0.0, 0.0] >>> cross_entropy_error(np.array(y), np.array(t)) 0.51082545709933802
訓練データすべての損失関数の平均
# データがひとつの場合と、データがバッチとしてまとめら れて入力される場合の両方のケースに対応
def cross_entropy_error(y, t):
# yが普通の配列だったとき(データ一個の出力だったとき)行列にする
if y.ndim == 1:
t = t.reshape(1, t.size)
y = y.reshape(1, y.size)
# 出力した画像全てをまとめて損失関数の総和を出す
# ちなみにy.shape[1]は出力層のニューロンの数
batch_size = y.shape[0]
return -np.sum(t * np.log(y)) / batch_sizeちなみにnp.sumや配列積は以下の例からわかるはず
>>> import numpy as np >>> x = np.arange(10).reshape(2,5) >>> y = np.arange(10).reshape(2,5) >>> x array([[0, 1, 2, 3, 4], [5, 6, 7, 8, 9]]) >>> x*y array([[ 0, 1, 4, 9, 16], [25, 36, 49, 64, 81]]) >>> np.sum(x*y) 285
教師データがラベルとして与えられたとき(one-hot 表現ではなく、「2」や 「7」といったラベルとして与えられたとき)、交差エントロピー誤差は次のように実
装することができます
def cross_entropy_error(y, t):
if y.ndim == 1:
t = t.reshape(1, t.size)
y = y.reshape(1, y.size)
batch_size = y.shape[0]
return -np.sum(np.log(y[np.arange(batch_size), t])) / batch_size実装のポイントは、one-hot 表現で t が 0 の要素は、交差エントロピー誤差 も 0 であるから、その計算は無視してもよい。
y[np.arange(batch_size), t] は 各画像行 正解ラベル列として
array([ 画像1の正解ラベル出力値, 画像2の正解ラベル出力値, 画像3の正解ラベル出力値, ... , 画像Nの正解ラベル出力値 ])
になっている。
認識精度が高くなるようなパラメータ を獲得したいので、「認識精度」を指標にすべきではないか?
認識精度は離散値であり、微分がほとんどの場所で 0 になってし まい、パラメータの更新(学習)ができなくなってしまう。
例えば、訓練データが100枚の時、精度は1%毎にしか変化できない
df(x)/dx = lim{(f(x + h) − f(x)) / h}
# 悪い実装例 def numerical_diff(f, x): h = 10e-50 return (f(x+h) - f(x)) / h
h には 10e-50(「0.00...1」の 0 が 50 個続く数)という小さな値を用いてます。しかし、これでは逆に丸め誤差になる。
丸め誤差とは, 小数の小さな範囲において数値が省略され ることで(たとえば、小数点第 8 位以下が省略されるといったこと)、最終的な計算 結果に誤差が生じる
>>> import numpy as np >>> np.float32(1e-50) 0.0
改善点1: 丸め誤差を避けるべく、微小な値 h として 10−4 を用いる。(10−4 程度 の値を用いれば、良い結果が得られることが分かってる。)
改善点2: (x, f(x)) (x+h, f(x+h)) の2点間の直線であり(前方差分)、接戦の傾きとは誤差があるので、誤差を減らす工夫とし て、(x + h) と (x − h) での関数 f の差分を計算する(中心差分)
def numerical_diff(f, x): h = 1e-4 # 0.0001 return (f(x+h) - f(x-h)) / (2*h)
上記のように微小な差分によって微分を求めることを数値微分
一方、数式の展開によって微分を「解析的に解く」とか「解析的に微分を求める」などと言います
(gradient_1d.py)
import numpy as np
import matplotlib.pylab as plt
def numerical_diff(f, x):
h = 1e-4 # 0.0001
return (f(x+h) - f(x-h)) / (2*h)
def function_1(x):
return 0.01*x**2 + 0.1*x
def tangent_line(f, x):
d = numerical_diff(f, x)
print(d)
# 接戦Y = dx + bより、以下はy切片bを表している
b = f(x) - d*x
# 傾きd y切片bの1次関数を返す関数を表している
return lambda t: d*t + b
x = np.arange(0.0, 20.0, 0.1)
y = function_1(x) # 関数1
plt.xlabel("x")
plt.ylabel("f(x)")
tf = tangent_line(function_1, 5)
y2 = tf(x) # 接戦
plt.plot(x, y)
plt.plot(x, y2)
plt.show()$ python gradient_1d.py 0.1999999999990898 # 真の微分は0.2なのでほとんど同じ値と見なすことができるぐらい小さな誤差
f(x0,x1)=x0² +x1²
def function_2(x): return x[0]**2 + x[1]**2 # または return np.sum(x**2)
x0 = 3、x1 = 4 のときの x0 に対する偏微分∂f / ∂x0
>>> def function_tmp1(x0): ... return x0*x0 + 4.0**2.0 ... >>> numerical_diff(function_tmp1, 3.0)6.00000000000378
x0 = 3、x1 = 4 のときの x1 に対する偏微分∂f / ∂x1
>>> def function_tmp2(x1): ... return 3.0**2.0 + x1*x1 ... >>> numerical_diff(function_tmp2, 4.0)7.999999999999119
(gradient_2d.py)
import numpy as np
import matplotlib.pylab as plt
from mpl_toolkits.mplot3d import Axes3D
# すべての変数の偏微分ベクトル(勾配)を求める
def _numerical_gradient_no_batch(f, x):
h = 1e-4 # 0.0001
grad = np.zeros_like(x) # xと形状が同じで値が全て0の配列
for idx in range(x.size):
tmp_val = x[idx]
x[idx] = float(tmp_val) + h
fxh1 = f(x) # f(x+h)
x[idx] = tmp_val - h
fxh2 = f(x) # f(x-h)
grad[idx] = (fxh1 - fxh2) / (2*h)
x[idx] = tmp_val # 値を元に戻す
return grad
# バッチ処理
def numerical_gradient(f, X):
if X.ndim == 1:
return _numerical_gradient_no_batch(f, X)
else:
grad = np.zeros_like(X)
for idx, x in enumerate(X):
grad[idx] = _numerical_gradient_no_batch(f, x)
return grad
def function_2(x):
if x.ndim == 1:
return np.sum(x**2)
else:
return np.sum(x**2, axis=1)
def tangent_line(f, x):
d = numerical_gradient(f, x)
print(d) # 微分結果
# 接戦のy切片d
b = f(x) - d*x
return lambda t: d*t + b
if __name__ == '__main__':
x0 = np.arange(-2, 2.5, 0.25)
x1 = np.arange(-2, 2.5, 0.25)
X, Y = np.meshgrid(x0, x1)
X = X.flatten()
Y = Y.flatten()
grad = numerical_gradient(function_2, np.array([X, Y]).T).T
plt.figure() # Figureインスタンスを作成
plt.quiver(X, Y, -grad[0], -grad[1], angles="xy",color="#666666") # 矢印(ベクトル)
plt.xlim([-2, 2])
plt.ylim([-2, 2])
plt.xlabel('x0')
plt.ylabel('x1')
plt.grid()
plt.draw()
plt.show()補足: enumerate
Pythonのenumerate()関数を使うと、forループの中でリスト(配列)などのイテラブルオブジェクトの要素と同時にインデックス番号(カウント、順番)を取得できる。
l = ['Alice', 'Bob', 'Charlie']
for i, name in enumerate(l):
print(i, name)
# 0 Alice
# 1 Bob
# 2 Charlie補足: np.ndarray.flatten 多次元配列を一次元配列
>>> x=np.arange(12).reshape(2,3,2) >>> x.flatten() array([ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11])
補足: meshgridについて
例えばx = [1,2,3,4,5,6,7,8,9]の配列をX軸y = [10,20,30,40,50]の配列をY軸として表を作る場合
y \ x | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 |
|---|---|---|---|---|---|---|---|---|---|
10 | |||||||||
20 | |||||||||
30 | |||||||||
40 | |||||||||
50 |
上のような表の各要素のx値とy値から各要素に入る値を求めることになる。
meshgrid() を使うと、予め上のような表(実際は行列)に各要素毎のx値とy値を埋めたものを生成してくれる。
import numpy as npx = [1,2,3,4,5,6,7,8,9]
y = [10,20,30,40,50]
X,Y = np.meshgrid(x,y)
print(X)
# => [[1 2 3 4 5 6 7 8 9]
# [1 2 3 4 5 6 7 8 9]
# [1 2 3 4 5 6 7 8 9]
# [1 2 3 4 5 6 7 8 9]
# [1 2 3 4 5 6 7 8 9]]
print(Y)
# => [[10 10 10 10 10 10 10 10 10]
# [20 20 20 20 20 20 20 20 20]
# [30 30 30 30 30 30 30 30 30]
# [40 40 40 40 40 40 40 40 40]
# [50 50 50 50 50 50 50 50 50]]
このような座標行列を予め作成しておくことより、
例えば
f(x,y) = x+yの値を求める場合
print(X+Y)
# => [[11 12 13 14 15 16 17 18 19]
# [21 22 23 24 25 26 27 28 29]
# [31 32 33 34 35 36 37 38 39]
# [41 42 43 44 45 46 47 48 49]
# [51 52 53 54 55 56 57 58 59]]
f(x,y) = x+2yの場合は
print(X+2*Y)
# => [[ 21 22 23 24 25 26 27 28 29]
# [ 41 42 43 44 45 46 47 48 49]
# [ 61 62 63 64 65 66 67 68 69]
# [ 81 82 83 84 85 86 87 88 89]
# [101 102 103 104 105 106 107 108 109]]
のようにシンプルに各座標値を求めることができます。
補足: plt.quiver ベクトル(矢印)について
plt.quiver(0.5,0.5,0.5,0.5) #(x,y,u,v) x,y-始点座標、u,v-ベクトルの向き
わかりやすかった参考文献 https://algorithm.joho.info/programming/python/matplotlib-quiver/
$ python gradient_2d.py
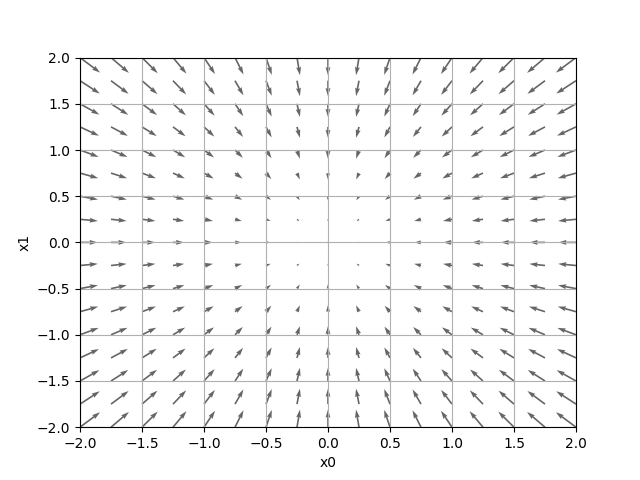
最小値を探す場合を勾配降下法(gradient descent method)、最大値を探す場合を勾配上昇法(gradient ascent method)と言います。
一般的に、ニューラルネットワーク(ディー プラーニング)の分野では、勾配法は「勾配降下法」として登場
η は学習率と呼ばれ、
学習率の値は、0.01 や 0.001 など、前もって何らかの値に決める必要がある。一般的に、大きすぎても小さすぎても、「良い場所」にたどり着くことができない。
# lr(learningrate)学習率, step_num 学習回数
def gradient_descent(f, init_x, lr=0.01, step_num=100):
x = init_x
for i in range(step_num):
grad = numerical_gradient(f, x)
x -= lr * grad
return x問: f(x0 , x1 ) = x0² + x1² の最小値を勾配法で求めよ
>>> def function_2(x): ... return x[0]**2 + x[1]**2... >>> init_x = np.array([-3.0, 4.0]) >>> gradient_descent(function_2, init_x=init_x, lr=0.1, step_num=100) array([ -6.11110793e-10, 8.14814391e-10])
最終的な結果は (-6.1e-10, 8.1e-10) となり、これはほとんど (0, 0)に近い。
真の最小値は (0, 0) なので、勾配法によって、ほぼ正 しい結果を得ることができた
(gradient_method.py)
import numpy as np
import matplotlib.pylab as plt
from gradient_2d import numerical_gradient
def gradient_descent(f, init_x, lr=0.01, step_num=100):
x = init_x
x_history = []
for i in range(step_num):
x_history.append( x.copy() ) # 配列に要素を追加
grad = numerical_gradient(f, x)
x -= lr * grad
return x, np.array(x_history)
def function_2(x):
return x[0]**2 + x[1]**2
init_x = np.array([-3.0, 4.0])
lr = 0.1
step_num = 20
x, x_history = gradient_descent(function_2, init_x, lr=lr, step_num=step_num)
#print("学習結果x")
#print(x)
#print("学習過程")
#print(x_history)
plt.plot( [-5, 5], [0,0], '--b') # blue markers with default shape
plt.plot( [0,0], [-5, 5], '--b')
plt.plot(x_history[:,0], x_history[:,1], 'o') # 'o' サークルマーカー
plt.xlim(-3.5, 3.5)
plt.ylim(-4.5, 4.5)
plt.xlabel("X0")
plt.ylabel("X1")
plt.show()補足: array.copy() オブジェクトをコピーする(値を参照ではなく全く別のものを複製)
・値渡しと参照渡し
参照渡し >>> x=[1,2,3] >>> y=x >>> y.apend(4) >>> y,x [1, 2, 3, 4], [1, 2, 3, 4]
上記は参照元と参照先がメモリを共有した状態であり、一方を変更したら両方変更が適用される
値渡し >>> x=[1,2,3] >>> y=x.copy() >>> y.append(4) >>> x,y ([1, 2, 3], [1, 2, 3, 4])
補足: matplotlib.pyplot.plotのオプションいついてはドキュメント参照
補足: array[:,0]やarray[:,1]について
[行:列]でスライスでき、省略した場合はすべてを指定したことになるので、[:, 0]は全ての行の0列目を取得することになります。省略せずに書くと2x2の配列の場合ならa[0:2, 0]となります。
>>> a = numpy.array([[0,1], [2, 3]]) >>> aarray([[0, 1], [2, 3]]) >>> a[:,0]array([0, 2]) >>>
$ python gradient_method.py
(gradient_simplenet.py)
import sys, os
sys.path.append(os.pardir)
import numpy as np from common.functions
from common.functions import softmax, cross_entropy_error
from common.gradient import numerical_gradient
class simpleNet:
def __init__(self):
self.W = np.random.randn(2,3) # ガウス分布で重み初期化
def predict(self, x):
return np.dot(x, self.W)
def loss(self, x, t):
z = self.predict(x)
y = softmax(z)
loss = cross_entropy_error(y, t)
return loss
x = np.array([0.6, 0.9])
t = np.array([0, 0, 1])
net = simpleNet()
f = lambda w: net.loss(x, t)
dW = numerical_gradient(f, net.W)
print(dW) # 重みの微分結果2 層ニューラルネットワークを、ひとつのクラスとして実装する
(two_layer_net.py)
import sys, os
sys.path.append(os.pardir) # 親ディレクトリのファイルをインポートするための設定
from common.functions import *
from common.gradient import numerical_gradient
class TwoLayerNet:
# 引数は、入力層のニューロンの数、隠れ層のニューロンの数、出力層のニューロンの数
def __init__(self, input_size, hidden_size, output_size, weight_init_std=0.01):
# 重みの初期化
self.params = {}
self.params['W1'] = weight_init_std * np.random.randn(input_size, hidden_size)
self.params['b1'] = np.zeros(hidden_size)
self.params['W2'] = weight_init_std * np.random.randn(hidden_size, output_size)
self.params['b2'] = np.zeros(output_size)
# 入力層のニューロンからニューラルネットワークの計算値を出力
def predict(self, x):
W1, W2 = self.params['W1'], self.params['W2']
b1, b2 = self.params['b1'], self.params['b2']
a1 = np.dot(x, W1) + b1
z1 = sigmoid(a1)
a2 = np.dot(z1, W2) + b2
y = softmax(a2)
return y
# 損失関数を求める x:入力データ, t:教師データ
def loss(self, x, t):
y = self.predict(x)
return cross_entropy_error(y, t)
# 認識精度(0~1)
def accuracy(self, x, t):
y = self.predict(x)
y = np.argmax(y, axis=1)
t = np.argmax(t, axis=1)
accuracy = np.sum(y == t) / float(x.shape[0])
return accuracy
# 重みパラメータに対する勾配を求める x:入力データ, t:教師データ
def numerical_gradient(self, x, t):
loss_W = lambda W: self.loss(x, t)
# 勾配を保持するディクショナリ変数
grads = {}
grads['W1'] = numerical_gradient(loss_W, self.params['W1'])
grads['b1'] = numerical_gradient(loss_W, self.params['b1'])
grads['W2'] = numerical_gradient(loss_W, self.params['W2'])
grads['b2'] = numerical_gradient(loss_W, self.params['b2'])
return grads
# 重みパラメータに対する勾配を求める numerical_gradient() の高速版!
def gradient(self, x, t):
W1, W2 = self.params['W1'], self.params['W2']
b1, b2 = self.params['b1'], self.params['b2']
grads = {}
batch_num = x.shape[0]
# forward
a1 = np.dot(x, W1) + b1
z1 = sigmoid(a1)
a2 = np.dot(z1, W2) + b2
y = softmax(a2)
# backward
dy = (y - t) / batch_num
grads['W2'] = np.dot(z1.T, dy) # 2層目の重みの勾配
grads['b2'] = np.sum(dy, axis=0) # 二層目のバイアスの勾配
dz1 = np.dot(dy, W2.T)
da1 = sigmoid_grad(a1) * dz1
grads['W1'] = np.dot(x.T, da1) # 1層目の重みの勾配
grads['b1'] = np.sum(da1, axis=0) # 一層目のバイアスの勾配
return gradsTwoLayerNet クラスを対象に、MNIST データセッ トを使って学習
(train_nueralnet.py)
# coding: utf-8
import sys, os
sys.path.append(os.pardir) # 親ディレクトリのファイルをインポートするための設定
import numpy as np
import matplotlib.pyplot as plt
from dataset.mnist import load_mnist
from two_layer_net import TwoLayerNet
# データの読み込み
(x_train, t_train), (x_test, t_test) = load_mnist(normalize=True, one_hot_label=True)
network = TwoLayerNet(input_size=784, hidden_size=50, output_size=10)
iters_num = 10000 # 勾配法による更新の回数(繰り返し回数)を適宜設定する
train_size = x_train.shape[0]
batch_size = 100
learning_rate = 0.1
train_loss_list = []
train_acc_list = []
test_acc_list = []
# 1エポックあたりの繰り返し数(訓練データの総数6万枚に相当する量を読み込んだ時全ての画像を見たと定め1エポック)
iter_per_epoch = max(train_size / batch_size, 1)
count=0
for i in range(iters_num):
# ミニバッチの取得
batch_mask = np.random.choice(train_size, batch_size) # train_size未満の自然数をbatch_size個セレクト
x_batch = x_train[batch_mask] # 例えば, x_train[[0,3]] だったら[x_train[0],x_train[3]] になる
t_batch = t_train[batch_mask]
# 勾配の計算
#grad = network.numerical_gradient(x_batch, t_batch)
grad = network.gradient(x_batch, t_batch)
# パラメータの更新(学習)
for key in ('W1', 'b1', 'W2', 'b2'):
network.params[key] -= learning_rate * grad[key]
# 損失関数を計算して配列にメモ
loss = network.loss(x_batch, t_batch)
train_loss_list.append(loss)
# 1エポック(バッチ)ごとに認識精度を計算 今回はiter_per_epochが600.0なので600回ごとに実行される
if i % iter_per_epoch == 0:
# print(i) => 0,600,1200, ... ,9600
train_acc = network.accuracy(x_train, t_train)
test_acc = network.accuracy(x_test, t_test)
train_acc_list.append(train_acc) # 精度をメモ
test_acc_list.append(test_acc) # 精度をメモ
print("train acc, test acc | " + str(train_acc) + ", " + str(test_acc))
# グラフの描画
markers = {'train': 'o', 'test': 's'}
x = np.arange(len(train_acc_list))
plt.plot(x, train_acc_list, label='train acc')
plt.plot(x, test_acc_list, label='test acc', linestyle='--')
plt.xlabel("epochs")
plt.ylabel("accuracy")
plt.ylim(0, 1.0)
plt.legend(loc='lower right')
plt.show()$ python train_neuralnet.py train acc, test acc | 0.09871666666666666, 0.098 train acc, test acc | 0.7983166666666667, 0.8035 train acc, test acc | 0.8775833333333334, 0.8794 train acc, test acc | 0.8989, 0.902 train acc, test acc | 0.9080333333333334, 0.9117 train acc, test acc | 0.9140666666666667, 0.9162 train acc, test acc | 0.91905, 0.9208 train acc, test acc | 0.92365, 0.9265 train acc, test acc | 0.9277333333333333, 0.9272 train acc, test acc | 0.9306166666666666, 0.9305 train acc, test acc | 0.9336333333333333, 0.9334 train acc, test acc | 0.9368166666666666, 0.9366 train acc, test acc | 0.93925, 0.9399 train acc, test acc | 0.9417333333333333, 0.9401 train acc, test acc | 0.9438833333333333, 0.942 train acc, test acc | 0.9452666666666667, 0.9444 train acc, test acc | 0.9467, 0.9455
学習をしていない初めは確率通りでほぼ10%であり、6万枚を16エポック96万枚学習した性能は94%である
訓練データとテストデータを使って評価した認識精度は両方とも向上している。
また、その 2 つの認識精度には差がないことが分かる(その 2 つの線はほぼ重なっている)。
そのため、今回の学習では過学習が起きていないことが 分かる。